seaborn.clustermap
译者:cancan233
seaborn.clustermap(data, pivot_kws=None, method='average', metric='euclidean', z_score=None, standard_scale=None, figsize=None, cbar_kws=None, row_cluster=True, col_cluster=True, row_linkage=None, col_linkage=None, row_colors=None, col_colors=None, mask=None, **kwargs)
将矩阵数据集绘制成分层聚类热图。
参数:data:2D array-like
用于聚类的矩形数据,不能包含 NA。
pivot_kws:字典,可选。
如果数据是整齐的数据框架,可以为 pivot 提供关键字参数以创建矩形数据框架。
method:字符串,可选。
用于计算聚类的链接方法。有关更多信息,请参阅文档 scipy.cluster.hierarchy.linkage https://docs.scipy.org/doc/scipy/reference/generated/scipy.cluster.hierarchy.linkage.html
metric:字符串,可选。
用于数据的距离度量。有关更多选项,请参阅 scipy.spatial.distance.pdist 文档。 https://docs.scipy.org/doc/scipy/reference/generated/scipy.spatial.distance.pdist.html 要对行和列使用不同的度量(或方法),您可以子集构造每个链接矩阵,并将它们提供为
{row, col}_linkage.
z_score:int 或 None,可选。
0(行)或 1(列)。是否计算行或列的 z 分数。Z 得分为 z = (x - mean)/std,因此每行(列)中的值将减去行(列)的平均值,然后除以行(列)的标准偏差。这可确保每行(列)的均值为 0,方差为 1.
standard_scale:int 或 None, 可选。
0(行)或 1(列)。是否标准化该维度,即每行或每列的含义,减去最小值并将每个维度除以其最大值。
figsize: 两个整数的元组, 可选。
要创建的图形的大小。
cbar_kws:字典, 可选。
要传递给
heatmap中的cbar_kws的关键字参数,例如向彩条添加标签。
{row,col}_cluster:布尔值, 可选。
如果为真,则对{rows, columns}进行聚类。
{row,col}_linkage:numpy.array, 可选。
行或列的预计算链接矩阵。有关特定格式,请参阅 scipy.cluster.hierarchy.linkage.
{row,col}_colors:list-like 或 pandas DataFrame/Series, 可选。
要为行或列标记的颜色列表。用于评估组内的样本是否聚集在一起。可以使用嵌套列表或 DataFrame 进行多种颜色级别的标注。如果以 DataFrame 或 Series 形式提供,则从 DataFrames 列名称或 Series 的名称中提取颜色标签。DataFrame/Series 颜色也通过索引与数据匹配,确保以正确的顺序绘制颜色。
mask:布尔数组或 DataFrame, 可选。
如果通过,数据将不会显示在
mask为真的单元格中。具有缺失值的单元格将自动被屏蔽。仅用于可视化,不用于计算。
kwargs:其他关键字参数。
所有其他关键字参数都传递给
sns.heatmap
返回值:clustergrid:ClusterGrid
ClusterGrid 实例。
注意点:
返回的对象有一个savefig方法,如果要保存图形对象而不剪切树形图,则应使用该方法。
要访问重新排序的行索引,请使用:clustergrid.dendrogram_row.reordered_in
列索引, 请使用: clustergrid.dendrogram_col.reordered_ind
范例
绘制聚类热图。
>>> import seaborn as sns; sns.set(color_codes=True)
>>> iris = sns.load_dataset("iris")
>>> species = iris.pop("species")
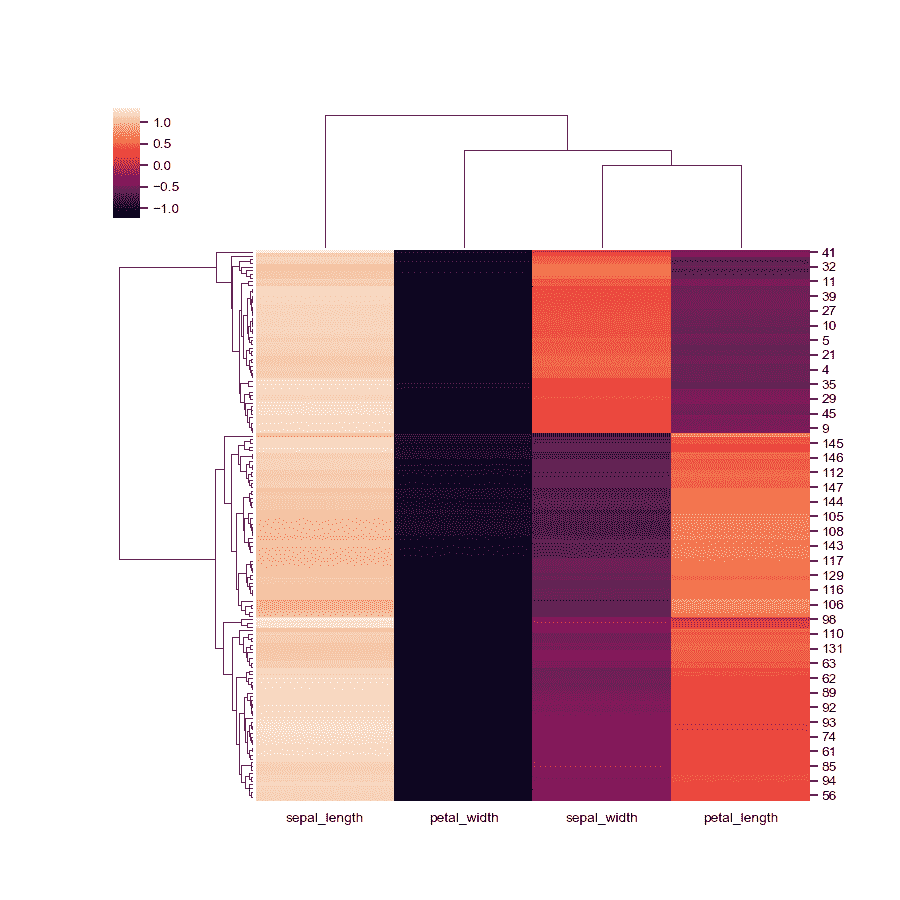
>>> g = sns.clustermap(iris)
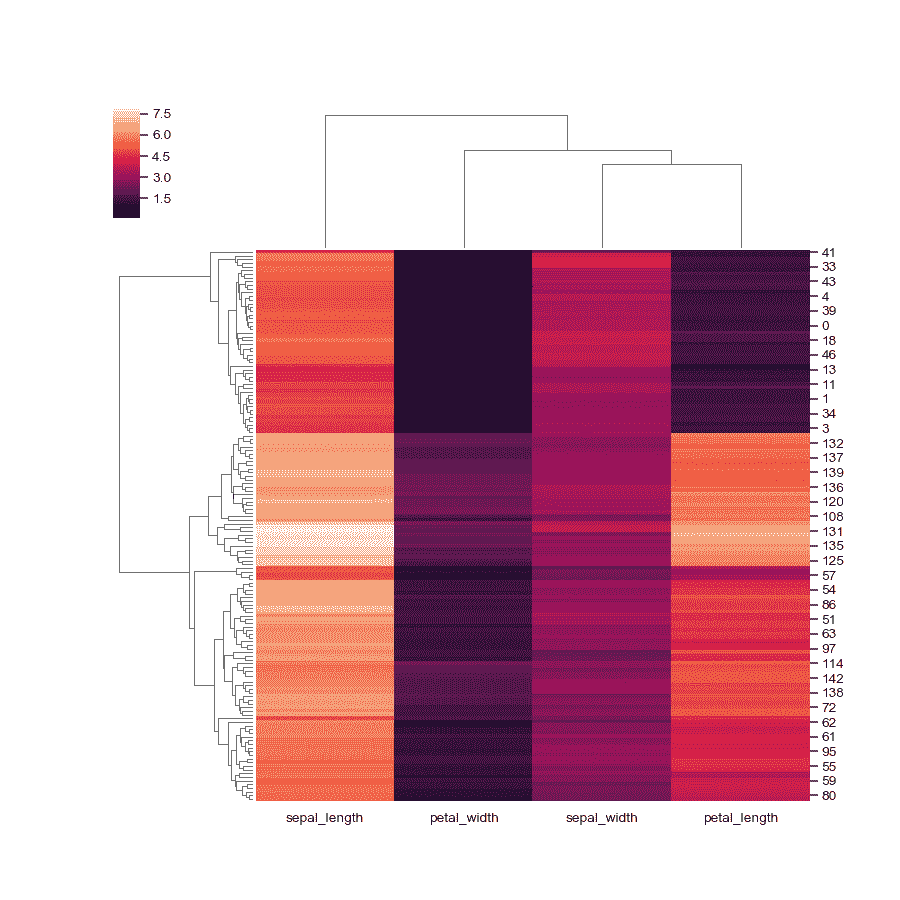
使用不同的相似性指标。
>>> g = sns.clustermap(iris, metric="correlation")
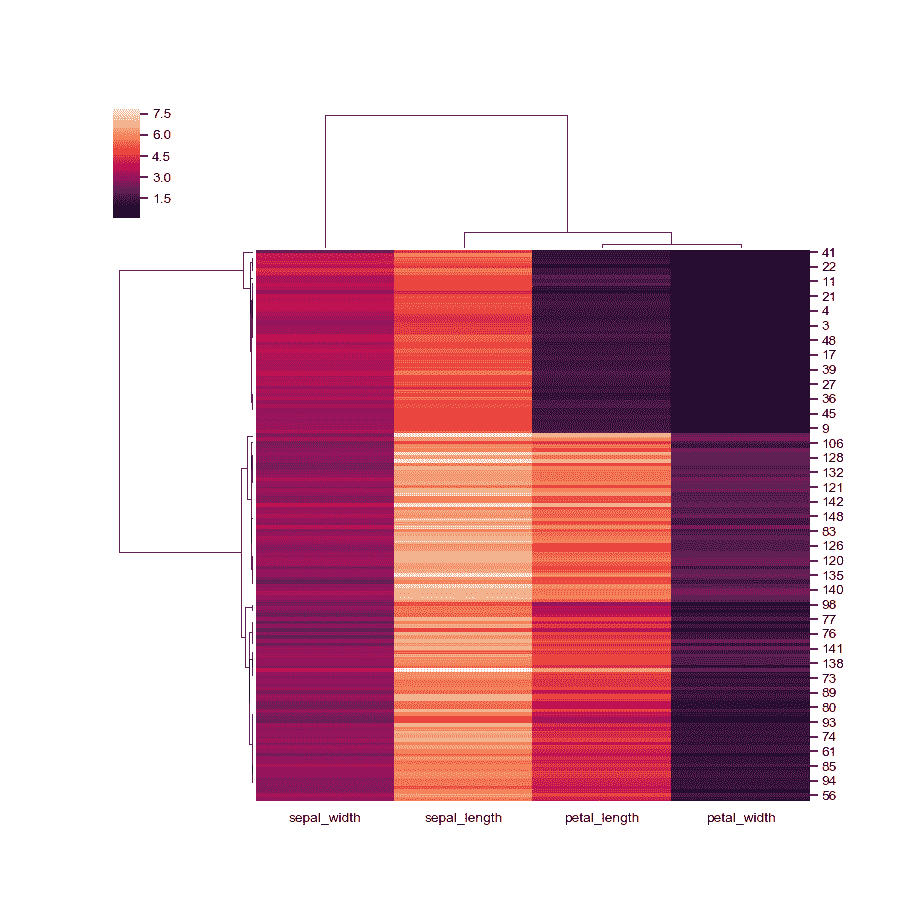
使用不同的聚类方法。
>>> g = sns.clustermap(iris, method="single")
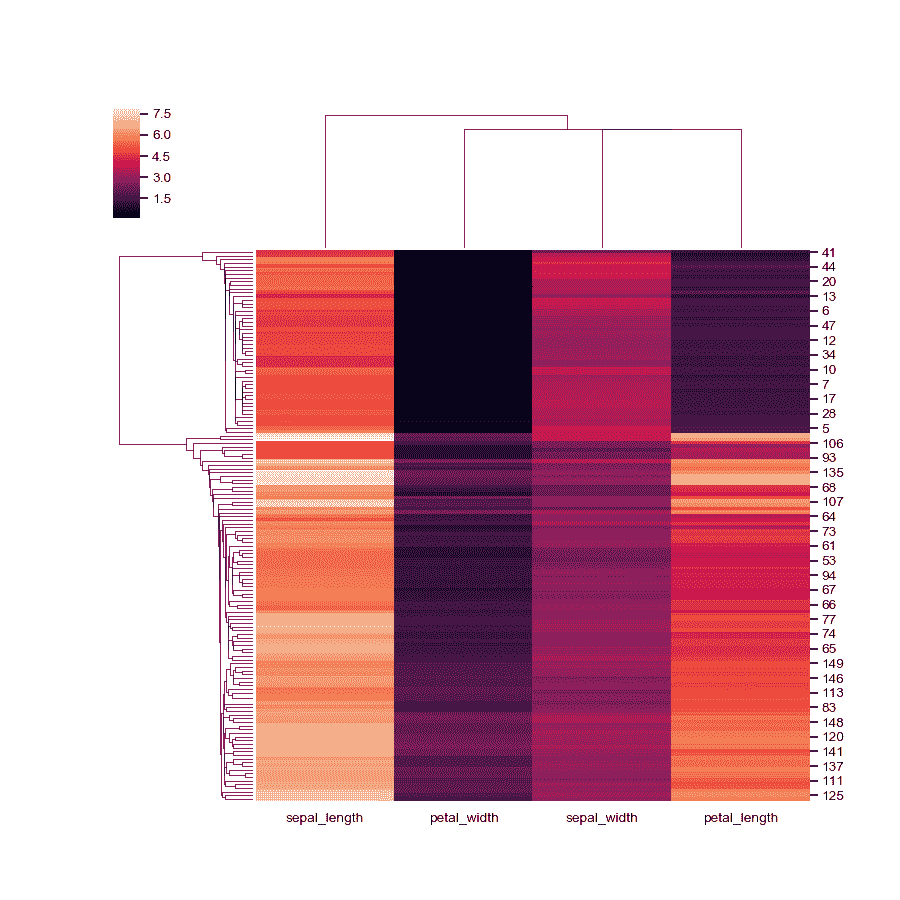
使用不同的色彩映射并忽略色彩映射限制中的异常值。
>>> g = sns.clustermap(iris, cmap="mako", robust=True)
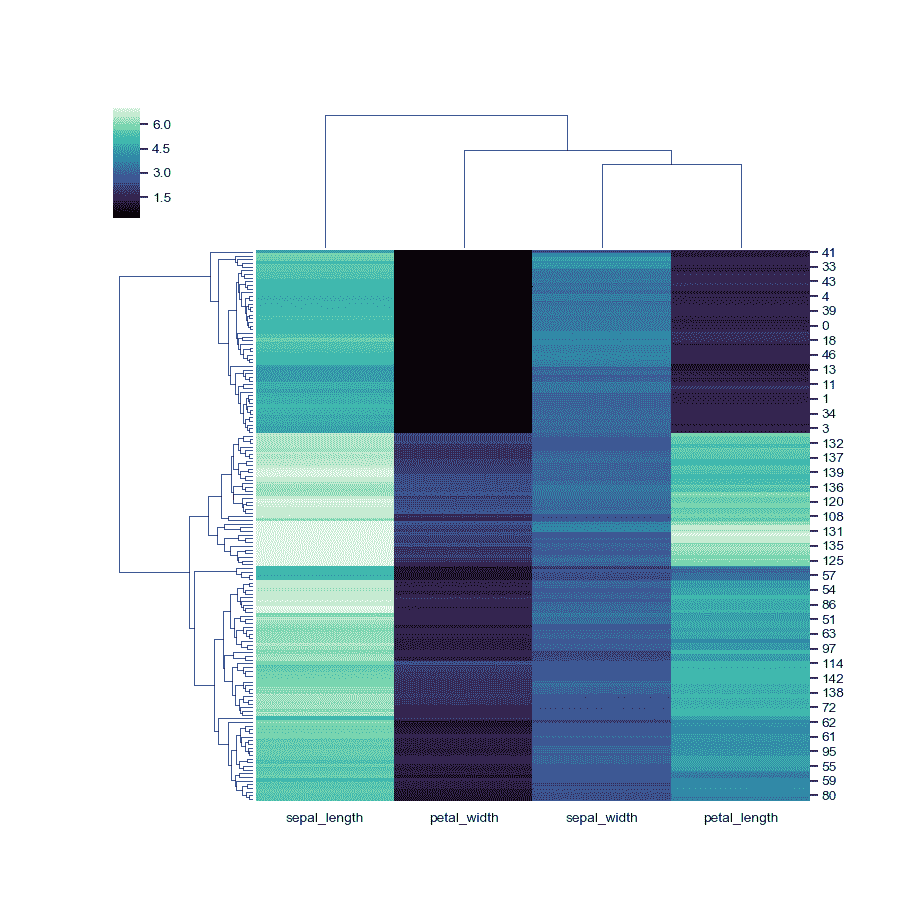
改变图的大小。
>>> g = sns.clustermap(iris, figsize=(6, 7))
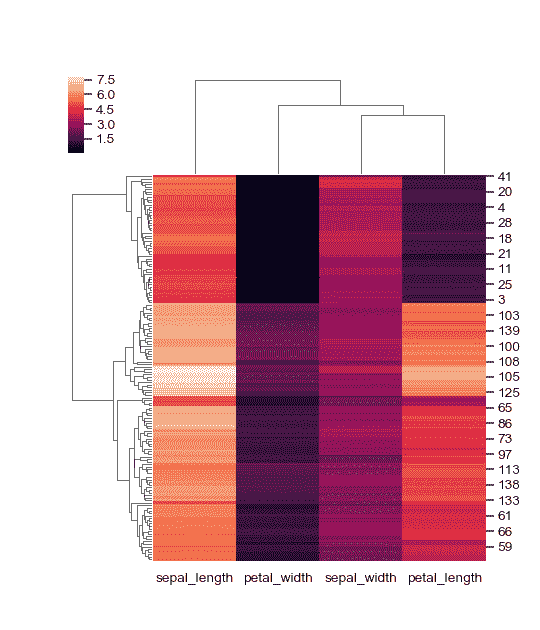
绘制其原始组织中的一个轴。
>>> g = sns.clustermap(iris, col_cluster=False)
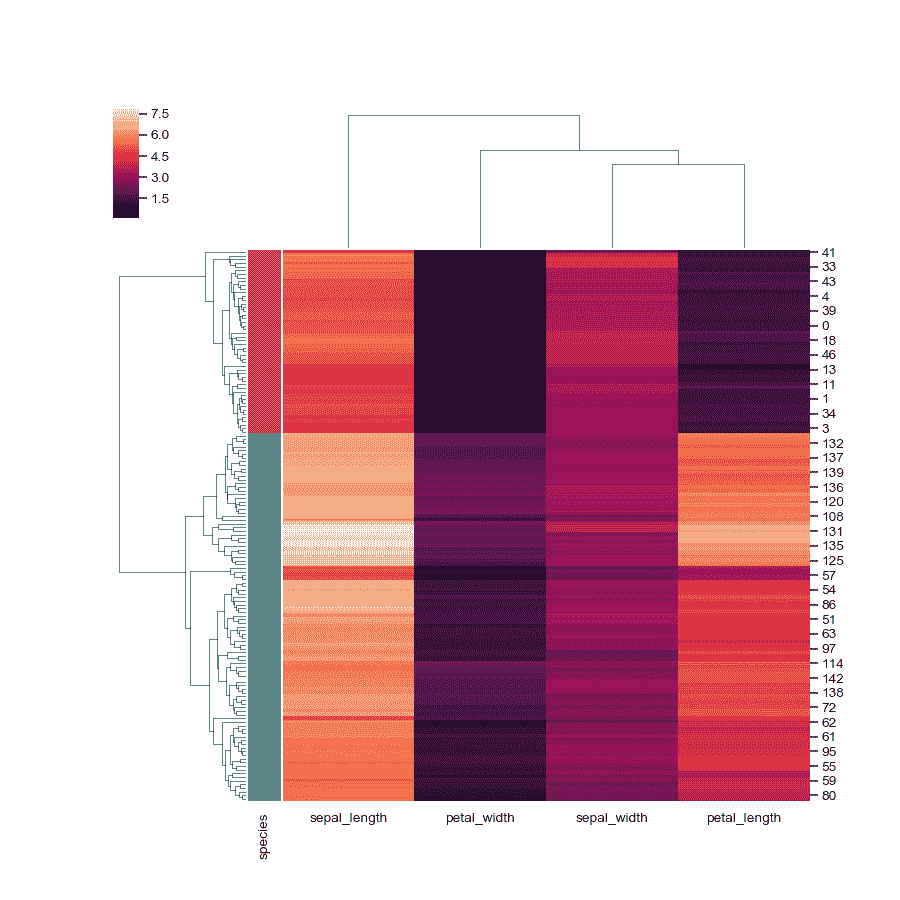
添加彩色标签。
>>> lut = dict(zip(species.unique(), "rbg"))
>>> row_colors = species.map(lut)
>>> g = sns.clustermap(iris, row_colors=row_colors)
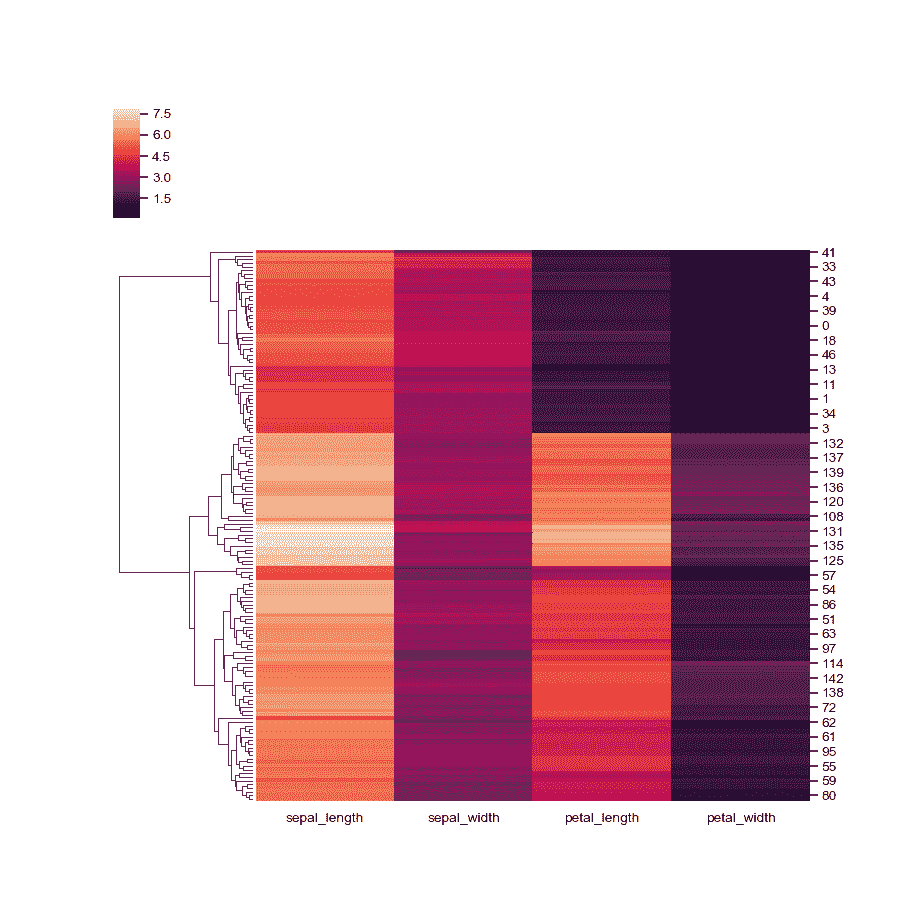
标准化列中的数据。
>>> g = sns.clustermap(iris, standard_scale=1)
正规化行内数据。
>>> g = sns.clustermap(iris, z_score=0)